В большинстве случаев в DS предпочитают работать с прямоугольными данными и на то есть масса причин и обоснований. Очень популярна тема парсинга и развертывания вложенных json. Часто дают тестовые задания на приведение к прямоугольному виду.
Но далеко не все задачи сводятся к data.frame и не обязательно весь DS сводить к ML. Есть масса задач, оперирующих, например, с графами или их частным случаем — деревьями. Можно использовать библиотеку igraph или аналоги и не думать о деталях. А можно попробовать чуть заглянуть внутрь алгоритмов.
На примере одной задачки посмотрим на работу с деревьями с альтернативной колокольни.
Все предыдущие публикации на habr.
Постановка задачи
Задачка в предельно упрощенном виде (сама по себе полная исходная задача куда сложнее), идет из тематики «process mining» и звучит так:
- Есть инстансы процессов и набор шагов, выполняемых в этих процессах.
- Система является конечным автоматом, набор процессов и состояний (шагов) ограничен и мал.
- Глядя на весь набор исторических данных и текущее состояние исполняемого инстанса процесса требуется предсказать следующий шаг.
Можно было бы эту задачу переформулировать в более наглядных трактовках, как то: наблюдение за проплывающими крокодилами; катание на автобусах без номера маршрута; гадание на ромашках и т.д.
Но пусть она останется в более серьезном виде.
Лог событий тоже предельно сжат с сохранением правильной временнОй последовательности и содержит только два идентификатора case_id и event, необходимых в контексте текущей задачи. Исходный лог
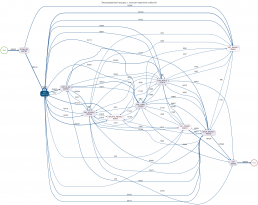
Визуально граф переходов (DFG) выглядит примерно следующей мешаниной:
fpath <- here::here("data", "scan_and_go_seq2_reduced")
raw_dt <- fs::path_ext_set(fpath, "zip") %>%
vroom::vroom(delim = ",", locale = locale("ru", encoding = "UTF-8")) %>%
select(timestamp, activity_id = event, case_id) %>%
setDT() %>%
unique() %>%
setorder(timestamp)План решения
Прежде чем хвататься за клавиатуру, прикинем на салфетке план решения задачи. Получается два пункта.
- Для каждого инстанса процесса перейдем от последовательности состояний к последовательности шагов по достижению состояния. Т.е. от состояний
A; B; Cперейдем кA; A-B; A-B-C. Дляk-го шага это называется префиксомkпроцесса. - Построим дерево префиксов и определим вероятности перехода по тем или иным веткам, опираясь на наблюдаемые количественные показатели.
- Для каждого незавершенного процесса будем считать его максимальный префикс и соотносить его с префиксами завершенных процессов. В случае совпадения мы можем предсказать следующее состояние в соответствии с построенным деревом префиксов.
Итак, появляются деревья, они же графы. Можно не раздумывая долго сразу брать библиотеку для работы с графами.
Но это не всегда оказывается легко, да и работа с графами в общем случае зачастую удобнее в объектной модели — затруднительно применять векторизацию.
Но мы же сидим в пространстве DS инструментов со своими сильными и слабыми сторонами. Оставим ООП для C++ и Java, попробуем альтернативный вариант. Для этого воспользуемся:
- редуцированным представлением графа (дерева) в виде
data.frameс двумя колонкамиfrom&to. Это таблица ребер, а в колонках будут записаны уникальные имена нод. - возможностями
data.tableв части работы по ссылкам (reference semantic).
Поскольку имена состояний достаточно длинные, префиксы будут несколько неудобны для промежуточного обозрения. Воспользуемся стандартным подходом — сочиним коротенький подстановочный словарик.
# для улучшение обозримости задачи построим словарик активностей
set.seed(1)
acdict_dt <- raw_dt[, .(activity_id = unique(activity_id))] %>%
.[, act_tag := head(unique(stri_rand_strings(.N * 5, 2, pattern = "[A-Z]")), .N)]
# и дополним исходный eventlog
dt <- merge(raw_dt, acdict_dt, by = "activity_id")
acdict_dtСобираем сводку по префиксам
Займемся пошаговой сверткой путей с подсчетом количественных показателей. В классическом варианте здесь могла бы быть рекурсия или bfs/dfs с последующей комбинаторной сборкой. Но пусть это рассказывается в учебных курсах, там это все очень хорошо разложено по полочкам.
Мы сделаем это другим образом, воспользуемся методом аналогий. Все многообразие трейсов даст на дерево (новогодняя гирлянда типа дождь/бахрома/занавес) у которого есть корень START (блок питания). Будем просто наматывать гирлянду на катушку, начиная от блока питания. По сути, отдаленный алгоритм bfs, но объединяем маркировку сразу со складыванием и расчетами.

Может возникнуть резонный вопрос, почему одним махом не склеить все трейсы? Ответ лежит на поверхности — нам нужные количественные показатели для каждого префикса цепочки чтобы иметь вероятности ветвлений.
Свертка путей
tic("Trace convolution")
accu_dt <- data.table(NULL)
# инициализируем цепочки, path -- уже пройденный путь
# для упрощения задачи отбросим сверхдлинные цепочки
step_dt <- dt[, N := .N, by = case_id] %>%
.[N <= quantile(.$N, probs = 0.8)] %>%
.[, .(from = "START", timestamp, to = act_tag, case_id)] %>%
setkey(case_id, timestamp)
while(nrow(step_dt) > 0){
tic("Another step")
# считаем статистику для текущих путей и вливаем в аккумулятор
accu_dt <- step_dt %>%
# поскольку все отсортировано по времени
# забираем только первую строчку для каждого кейса,
.[, head(.SD, 1), by = case_id] %>%
.[, .(N = .N, rel = .N /nrow(.)), by = .(from, to)] %>%
.[, to := stri_c(from, to, sep = "-")] %>%
{rbindlist(list(accu_dt, .))}
# втягиваем текущее ребро в путь и переходим к следующему
step_dt[, `:=`(from = stri_c(from, head(to, 1), sep = "-"),
to = shift(to, type = "lead")), by = case_id]
# подчищаем хвосты цепочек
step_dt %<>% na.omit()
toc()
}
toc()Вариант реализации не сильно шустрый, но это вполне поправимо. Можно оптимизировать выборки и склейки, добавить преаллокацию памяти и сделать сдвиг однократным за счет буферных зон, но конкретно здесь и сейчас это некритично. Первичен простой и компактный код.
Итак, эту часть задачи удалось решить путем простых манипуляций с data.frame без привлечения пакетов по работе с графами (но держа алгоримты в голове).
Визуализируем
Дерево получилось разветвленное, для просмотра исключим «шумовые» ветвления. Для начала воспользуемся штатными механизмами графовых библиотек. Сначала достанем igraph.
accu_dt %>%
setorder(-N) %>%
.[, ratio := N / uniqueN(dt$case_id)]
ig <- accu_dt[ratio > 0.01, .(from, to)] %>%
graph_from_data_frame(directed = TRUE)
plot(ig, vertex.size = 3,
vertex.label.cex = .8,
vertex.label.dist = runif(length(V(ig)), -.7, .7),
edge.arrow.size = 0.5,
layout = layout.reingold.tilford)Мда, применительно к нашему набору данных визуализация не очень. И это еще при условии, что названия событий заменены на словарные сокращения. Тут нужно более информативное размещение узлов, хотя бы без перекрытий.
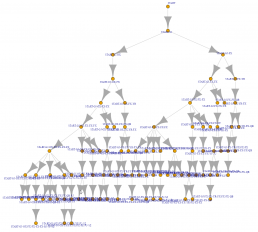
В R есть весьма функциональный пакет data.tree по работе именно с деревьями. Получаем вполне удобное и читаемое представление.
tree_dt <- accu_dt %>%
filter(ratio > 0.01) %>%
select(from, to)
print(FromDataFrameNetwork(tree_dt))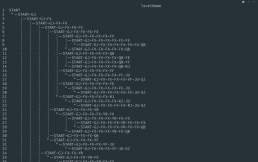
Но, хотелось бы отрисовать это в графике. Попробуем альтернативные варианты.
Можно использовать различные js виджеты, например, D3.
networkD3::simpleNetwork(tree_dt, fontSize = 12)Но тут возникает сложность. Дерево — частный случай графа, но для визуализации именно дерева надо готовить входные данные в структуре parent-child. Просто подать на вход таблицу ребер не получится.
Опять дело пахнет рекурсией, объектами, обходами и пр. Однако, попробуем решить это другими способами. Опять же, data.table и работа по ссылкам. Для визуализации дерева достанем еще один популярный виджен echarts. Непосредственно для него необходимо будет готовить подобную структуру входных данных:
tibble(
name = "earth",
# 1st level
children = list(
tibble(
name = c("land", "ocean"),
# 2nd level
children = list(
tibble(name = c("forest", "river")),
# 3rd level
tibble(
name = c("fish", "kelp"),
children = list(
tibble(
name = c("shark", "tuna")
),
# 4th level
NULL # kelp
))))))План свертки будет примерно аналогичен первой задаче, только сворачивать мы будем теперь не от блока питания, а от лампочек:
- расчитываем расстояния нод от корня;
- сворачиваем одним махом самые дальние ноды;
- движем вплоть до достижения корня.
Никаких рекурсий, никаких прогулок по ребрам. Определили конец и катим рулон. По своей сути, это обратная задача к мегапопулярному запросу «как мне развернуть вложенный json».

Свертка нод
# глубина дерева может быть любой, сворачивать придется с конца
tree_dt <- copy(accu_dt) %>%
.[ratio > 0.01, .(from, to, children = list(NULL))] %>%
# дополним расстояниями от корня
.[from == "START", dist := 0L]
# посчитаем расстояния нод от корня
while(tree_dt[is.na(dist), .N] > 0){
max_dist <- tree_dt[, max(dist, na.rm = TRUE)]
nodes <- tree_dt[dist == max_dist, unique(to)]
tree_dt[from %chin% nodes, dist := max_dist + 1L]
}
for(ii in max(tree_dt$dist):0) {
nested_dt <- tree_dt[dist == ii, .(from, name = to, children)] %>%
tidyfst::nest_dt(from, .name = "children")
# теперь делаем возвратную точечную вставку сверток
tree_dt[nested_dt, children := i.children, on = c(to = "from")]
}
# в последнем цикле нам вставка уже не нужна
nested_dt %>%
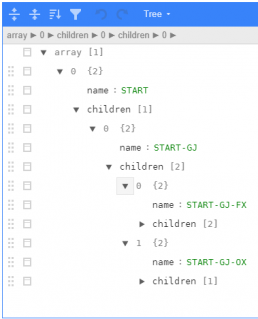
setnames("from", "name")Можно взглянуть на получившуюся структуру. Смотреть удобнее в интерактивных json виджетах.
jsonlite::toJSON(nested_dt) %>%
listviewer::jsonedit()Теперь можем построить интерактивный виджет echarts tree. Удобно и читаемо.
nested_dt |>
e_charts() |>
e_tree(initialTreeDepth = 10, label = list(offset = c(0, -11)))Заключение
Текущая публикация — просто очередная иллюстрация, как можно иначе взглянуть на решение «типовых задач». В DS инструментов чуть побольше, чем топор и пила.
- Компактно? Да.
- Быстро? Да.
- Понятно? Да.